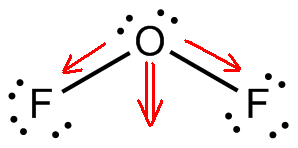
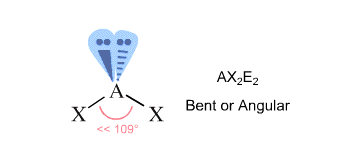Sauerstoffdifluorid, #“OF“_2#, ist ein polares Molekül, weil es eine gekrümmte Molekülgeometrie besitzt.
Diese Molekülgeometrie sorgt dafür, dass sich die Dipolmomente der Sauerstoff-Fluorid-Bindungen nicht gegenseitig aufheben und ein unpolares Molekül entsteht.
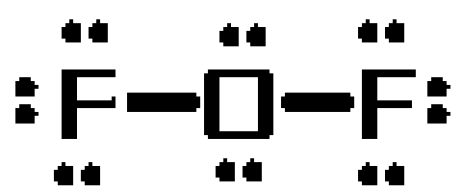
Um zu sehen, warum dies der Fall ist, zeichnen Sie die Lewis-Struktur des Moleküls. Das Molekül hat insgesamt #20# Valenzelektronen
- #6# vom Sauerstoffatom
- #7# von jedem der beiden Fluoratome
Das Sauerstoffatom übernimmt die Rolle des Zentralatoms und bildet Einfachbindungen mit den beiden Fluoridatomen. Auf diese Bindungen entfallen #4# der #20# Valenzelektronen des Moleküls.
Die resultierenden #16# Elektronen werden als einsame Paare platziert
- drei einsame Paare an jedem Fluoratom
- zwei einsame Paare am Sauerstoffatom
Nun, es ist sehr wichtig zu erkennen, dass die Lewis-Strukturen nicht dazu gedacht sind, die molekulare Geometrie zu vermitteln!
Um die Geometrie des Moleküls zu finden, zählen Sie die Bereiche der Elektronendichte, die das Zentralatom umgeben – diese geben Ihnen die sterische Zahl des Atoms.
Regionen der Elektronendichte sind Bindungen zu anderen Atomen (hier zählen Einfach-, Doppel- oder Dreifachbindungen als eine Region) und einsame Elektronenpaare.
In Ihrem Fall ist das zentrale Sauerstoffatom an zwei andere Atome gebunden und ist von zwei einsamen Elektronenpaaren umgeben #-># es hat eine sterische Zahl gleich #4#.
Nach der VSEPR-Theorie entspricht dies einer #“AX“_2 „E“_2# Molekülgeometrie, die für ein gebogenes Molekül charakteristisch ist.
Nun, der Unterschied in der Elektronegativität zwischen Fluor und Sauerstoff sorgt dafür, dass die beiden #“O“-„F „#-Bindungen polar sind. Durch die gebogene Molekülgeometrie werden sich die beiden Dipolmomente addieren.
Das Ergebnis wird die Bildung eines permanenten Dipolmoments sein, und somit ein polares Molekül